Diferencia entre revisiones de «Biología molecular»
Esta obra contiene una traducción derivada del articulo respectivo de Wikipedia en inglés, concretamente la versión válida al dia de la traducción, publicada por sus editores bajo la Licencia de documentación libre de GNU y la Licencia Creative Commons Atribución-CompartirIgual 3.0 Unported. |
Deshecha la edición 161597432 de 187.194.216.68 (disc.) Etiqueta: Deshecho |
||
| (No se muestran 23 ediciones intermedias de 14 usuarios) | |||
| Línea 19: | Línea 19: | ||
Los métodos que emplea esta nueva ciencia son fundamentalmente los mismos que la Biofísica, Bioquímica, y Biología. Utiliza los análisis químicos, cualitativo y cuantitativo, los conocimientos de la Química orgánica, la Biología de microorganismos y de virus, etc., pero revisten especial importancia los nuevos microanalíticos tanto físicos como químicos. Merecen destacarse la [[microscopía electrónica]], que permite resoluciones que alcanzan los 10 Amstrongs; la [[difracción de rayos X]], que determina la estructura y disposición espacial de los átomos de las macromoléculas; la ultracentrifugación diferencial, tanto analítica como preparativa, que permite separaciones antes imposibles; la [[cromatografía]] de gases, y, en fase líquida, la espectrografía de infrarrojos, la Química con isótopos trazadores, la espectrometría de masas, etc.{{Cita requerida}} |
Los métodos que emplea esta nueva ciencia son fundamentalmente los mismos que la Biofísica, Bioquímica, y Biología. Utiliza los análisis químicos, cualitativo y cuantitativo, los conocimientos de la Química orgánica, la Biología de microorganismos y de virus, etc., pero revisten especial importancia los nuevos microanalíticos tanto físicos como químicos. Merecen destacarse la [[microscopía electrónica]], que permite resoluciones que alcanzan los 10 Amstrongs; la [[difracción de rayos X]], que determina la estructura y disposición espacial de los átomos de las macromoléculas; la ultracentrifugación diferencial, tanto analítica como preparativa, que permite separaciones antes imposibles; la [[cromatografía]] de gases, y, en fase líquida, la espectrografía de infrarrojos, la Química con isótopos trazadores, la espectrometría de masas, etc.{{Cita requerida}} |
||
==Técnicas== |
|||
===Clonación molecular=== |
===Clonación molecular=== |
||
[[File:Transduction image.pdf|thumb|Imagen de transducción.]] |
[[File:Transduction image.pdf|thumb|Imagen de transducción.]] |
||
La clonación molecular se utiliza para aislar y luego transferir una secuencia de ADN de interés a un vector plásmido.<ref>{{Cite web|title=Fundamentos de la clonación molecular - Pasado, presente y futuro {{!}} NEB|url=https://www.neb.com/tools-and-resources/feature-articles/foundations-of-molecular-cloning-past-present-and-future|access-date=2021-11-25|website=www.neb.com}}</ref> Esta tecnología de ADN recombinante se desarrolló por primera vez en la década de 1960.<ref>{{Cite web|title=Fundamentos de la clonación molecular - Pasado, presente y futuro {{!}} NEB|url=https://www.neb.com/tools-and-resources/feature-articles/foundations-of-molecular-cloning-past-present-and-future|access-date=2021-11-04|website=www.neb.com}}</ref> En esta técnica, una secuencia de [[ADN]] que codifica una proteína de interés se [[clonación|clona]] mediante [[reacción en cadena de la polimerasa]] (PCR), y/o [[enzima de restricción]] |
La clonación molecular se utiliza para aislar y luego transferir una secuencia de ADN de interés a un vector plásmido.<ref>{{Cite web|title=Fundamentos de la clonación molecular - Pasado, presente y futuro {{!}} NEB|url=https://www.neb.com/tools-and-resources/feature-articles/foundations-of-molecular-cloning-past-present-and-future|access-date=2021-11-25|website=www.neb.com}}</ref> Esta tecnología de ADN recombinante se desarrolló por primera vez en la década de 1960.<ref>{{Cite web|title=Fundamentos de la clonación molecular - Pasado, presente y futuro {{!}} NEB|url=https://www.neb.com/tools-and-resources/feature-articles/foundations-of-molecular-cloning-past-present-and-future|access-date=2021-11-04|website=www.neb.com}}</ref> En esta técnica, una secuencia de [[ADN]] que codifica una proteína de interés se [[clonación|clona]] mediante [[reacción en cadena de la polimerasa]] (PCR), y/o [[enzima de restricción]], en un [[plásmido]] ([[vector de expresión]]). El plásmido vector suele tener al menos 3 características distintivas: un origen de replicación, un [[sitio de clonación múltiple|Sitio de Clonación Múltiple]] (SCM) y un marcador selectivo (normalmente [[resistencia a antibióticos]]). Además, aguas arriba del SCM se encuentran la [[Promotor (genética)|región promotora]] y la [[Transcripción genética|transcripción]], que regulan la expresión del gen clonado. |
||
Este plásmido puede insertarse en células bacterianas o animales. La introducción de ADN en células bacterianas puede realizarse mediante [[transformación (genética)|transformación]] por captación de ADN desnudo, [[conjugación bacteriana|conjugación]] por contacto célula-célula o mediante [[Transducción (genética)|transducción]] por vector viral. La introducción de ADN en células [[ |
Este plásmido puede insertarse en células bacterianas o animales. La introducción de ADN en células bacterianas puede realizarse mediante [[transformación (genética)|transformación]] por captación de ADN desnudo, [[conjugación bacteriana|conjugación]] por contacto célula-célula o mediante [[Transducción (genética)|transducción]] por vector viral. La introducción de ADN en células [[eucariotas]], como las células animales, por medios físicos o químicos se denomina [[transfección]]. Existen diversas técnicas de transfección, como la transfección con fosfato cálcico, la [[electroporación]], la [[microinyección]] y la [[transfección|transfección con liposomas]]. El plásmido puede integrarse en el [[genoma]], dando lugar a una transfección estable, o puede permanecer independiente del genoma y expresarse temporalmente, lo que se denomina transfección transitoria.<ref name="cell">{{cite book|last1=Alberts|first1=Bruce|last2=Johnson|first2=Alexander|last3=Lewis|first3=Julian|last4=Raff|first4=Martin|last5=Roberts|first5=Keith|last6=Walter|first6=Peter |title=Aislamiento, Clonación y secuenciación del ADN: //www. ncbi.nlm.nih.gov/books/NBK26837/|access-date=31 de diciembre de 2016|language=es}}</ref><ref>{{cite book|last1=Lessard|first1=Juliane C.|title=Métodos de laboratorio en enzimología: DNA|chapter=Molecular cloning|journal=Methods in Enzymology|date=2013-01-01|volume=529|pages=85–98|doi=10.1016/B978-0-12-418687-3.00007-0|pmid=24011038|issn=1557-7988|isbn=978-0-12-418687-3}}</ref> |
||
El ADN que codifica una proteína de interés se encuentra ahora dentro de una célula y la [[proteína]] puede expresarse. Existen diversos sistemas, como promotores inducibles y factores específicos de señalización celular, que ayudan a expresar la proteína de interés a niveles elevados. A continuación, se pueden extraer grandes cantidades de una proteína de la célula bacteriana o eucariota. La proteína puede someterse a pruebas de actividad enzimática en diversas situaciones, puede cristalizarse para estudiar su [[estructura terciaria]] o, en la industria farmacéutica, puede estudiarse la actividad de nuevos fármacos contra la proteína.<ref>{{cite book|last1=Kokate |first1=Chandrakant|last2=Jalalpure|first2=Sunil S. |last3=Hurakadle|first3=Pramod J. |title=Libro de texto de Biotecnología Farmacéutica|department=Clonación de expresiones|url=https://books.google.com/books?id=p70UCwAAQBAJ&pg=PA125|date=2016|publisher=Elsevier|page=125|access-date=2019-07-08|isbn=9788131239872}}</ref> |
El ADN que codifica una proteína de interés se encuentra ahora dentro de una célula y la [[proteína]] puede expresarse. Existen diversos sistemas, como promotores inducibles y factores específicos de señalización celular, que ayudan a expresar la proteína de interés a niveles elevados. A continuación, se pueden extraer grandes cantidades de una proteína de la célula bacteriana o eucariota. La proteína puede someterse a pruebas de actividad enzimática en diversas situaciones, puede cristalizarse para estudiar su [[estructura terciaria]] o, en la industria farmacéutica, puede estudiarse la actividad de nuevos fármacos contra la proteína.<ref>{{cite book|last1=Kokate |first1=Chandrakant|last2=Jalalpure|first2=Sunil S. |last3=Hurakadle|first3=Pramod J. |title=Libro de texto de Biotecnología Farmacéutica|department=Clonación de expresiones|url=https://books.google.com/books?id=p70UCwAAQBAJ&pg=PA125|date=2016|publisher=Elsevier|page=125|access-date=2019-07-08|isbn=9788131239872}}</ref> |
||
===Reacción en cadena de la polimerasa=== |
===Reacción en cadena de la polimerasa=== |
||
La reacción en cadena de la polimerasa (PCR) es una técnica extremadamente versátil para copiar ADN. En pocas palabras, la PCR permite copiar o modificar una [[secuenciación de ADN|secuencia de ADN]] específica de formas predeterminadas. La reacción es extremadamente potente y, en condiciones perfectas, podría amplificar una molécula de ADN hasta convertirla en 1.070 millones de moléculas en menos de dos horas. La PCR tiene muchas aplicaciones, como el estudio de la expresión génica, la detección de microorganismos patógenos, la detección de mutaciones genéticas y la introducción de mutaciones en el ADN.<ref>{{Cite journal|last=Lenstra|first=J. A.|fecha=Julio de 1995|title=Las aplicaciones de la reacción en cadena de la polimerasa en las ciencias de la vida|url=https://pubmed.ncbi.nlm.nih. |
La reacción en cadena de la polimerasa (PCR) es una técnica extremadamente versátil para copiar ADN. En pocas palabras, la PCR permite copiar o modificar una [[secuenciación de ADN|secuencia de ADN]] específica de formas predeterminadas. La reacción es extremadamente potente y, en condiciones perfectas, podría amplificar una molécula de ADN hasta convertirla en 1.070 millones de moléculas en menos de dos horas. La PCR tiene muchas aplicaciones, como el estudio de la expresión génica, la detección de microorganismos patógenos, la detección de mutaciones genéticas y la introducción de mutaciones en el ADN.<ref>{{Cite journal|last=Lenstra|first=J. A.|fecha=Julio de 1995|title=Las aplicaciones de la reacción en cadena de la polimerasa en las ciencias de la vida|url=https://pubmed.ncbi.nlm.nih.gov/7580841/|journal=Cellular and Molecular Biology (Noisy-Le-Grand, Francia)|volume=41|issue=5|pages=603-614|issn=0145-5680|pmid=7580841}}</ref> La técnica PCR puede utilizarse para introducir [[Sitio de restricción|sitios de enzimas de restricción]] en los extremos de las moléculas de ADN, o para mutar bases particulares del ADN, este último es un método conocido como [[mutagénesis dirigida al sitio]]. La PCR también puede utilizarse para determinar si un fragmento concreto de ADN se encuentra en una [[biblioteca de ADNc]]. La PCR tiene muchas variaciones, como la PCR de transcripción inversa ([[RT-PCR]]) para la amplificación de ARN y, más recientemente, la [[PCR cuantitativa]] que permite la medición cuantitativa de moléculas de ADN o ARN.<ref>{{cite web|title=Reacción en cadena de la polimerasa (PCR)|url=https://www.ncbi.nlm.nih.gov/probe/docs/techpcr/| work = National Center for Biotechnology Information | publisher = U.S. National Library of Medicine |access-date=31 de diciembre de 2016}}</ref><ref>{{cite web|title=Hoja informativa sobre la reacción en cadena de la polimerasa (PCR)|url=https://www.genome.gov/10000207/polymerase-chain-reaction-pcr-fact-sheet/|website=Instituto Nacional de Investigación del Genoma Humano (NHGRI)|access-date=31 de diciembre de 2016}}</ref> |
||
[[File:Two percent Agarose Gel in Borate Buffer cast in a Gel Tray (Front, angled).jpg||thumb|Gel de agarosa al dos por ciento en tampón borato salino moldeado en una bandeja de gel.]] |
[[File:Two percent Agarose Gel in Borate Buffer cast in a Gel Tray (Front, angled).jpg||thumb|Gel de agarosa al dos por ciento en tampón borato salino moldeado en una bandeja de gel.]] |
||
===Electroforesis en gel=== |
|||
[[File:SDS-PAGE.jpg|thumb|125px|SDS-PAGE|izquierda]] |
|||
La electroforesis en gel es una técnica que separa moléculas por su tamaño utilizando un gel de agarosa o poliacrilamida.<ref name="Lee-2012">{{Cite journal|last1=Lee|first1=Pei Yun|last2=Costumbrado|first2=John|last3=Hsu|first3=Chih-Yuan|last4=Kim|first4=Yong Hoon|date=2012- 04-20|title=Electroforesis en gel de agarosa para la separación de fragmentos de ADN|journal=Journal of Visualized Experiments|issue=62|pages=3923|doi=10.3791/3923|issn=1940-087X|pmc=4846332|pmid=22546956}}</ref> Esta técnica es una de las principales herramientas de la biología molecular. El principio básico es que los fragmentos de ADN se pueden separar aplicando una corriente eléctrica a través del gel - debido a que la columna vertebral del ADN contiene grupos fosfato cargados negativamente, el ADN migrará a través del gel de agarosa hacia el extremo positivo de la corriente.<ref name="Lee-2012" /> Las proteínas también pueden separarse en función del tamaño utilizando un gel [[SDS-PAGE]], o en función del tamaño y su [[carga eléctrica]] utilizando lo que se conoce como [[Electroforesis en gel bidimensional|Electroforesis en gel 2D]].<ref>{{cite journal | vauthors = Lee PY, Costumbrado J, Hsu CY, Kim YH | title = Electroforesis en gel de agarosa para la separación de fragmentos de ADN | journal = Journal of Visualized Experiments | issue = 62 | date = April 2012 | pmid = 22546956 | pmc = 4846332 | doi = 10.3791/3923 }}</ref>. |
|||
[[File:Coomassie blue stained gel.png|thumb|160x160px|Proteínas teñidas en un [[gel PAGE]] usando colorante azul de Coomassie.]] |
|||
== Contenido == |
== Contenido == |
||
| Línea 59: | Línea 65: | ||
== Bibliografía == |
== Bibliografía == |
||
* C. PETIT y G. PRÉVOST, Genetique et évolution, «Journal of Molecular Biologyu, Londres 1967; C. A. VILLE, Biología, México 1966. |
* C. PETIT y G. PRÉVOST, Genetique et évolution, «Journal of Molecular Biologyu, Londres 1967; C. A. VILLE, Biología, México 1966. |
||
* {{Enlace roto|1=[http://www.ornl.gov/hgmis/home.shtml Genome Glossary. Información sobre proyecto de genoma humano] |2=http://www.ornl.gov/hgmis/home.shtml |
* {{Enlace roto|1=[http://www.ornl.gov/hgmis/home.shtml Genome Glossary. Información sobre proyecto de genoma humano] |2=http://www.ornl.gov/hgmis/home.shtml }} {{en}} (accedido el 15 de junio de 2009) |
||
== Enlaces externos == |
== Enlaces externos == |
||
Revisión actual - 10:32 26 ago 2024

La biología molecular es la rama de la biología que tiene como objetivo el estudio de los procesos que se desarrollan en los seres vivos desde un punto de vista molecular. En su sentido moderno, la biología molecular pretende explicar los fenómenos de la vida a partir de sus propiedades macromoleculares. Dos macromoléculas en particular son su objeto de estudio:
- Los ácidos nucleicos, entre los cuales el más utilizado es el ácido desoxirribonucleico (ADN), el componente de los genes.
- Las proteínas, que son los agentes activos de los organismos vivos.
Dentro del Proyecto Genoma Humano puede encontrarse la siguiente definición sobre la biología molecular: El estudio de la estructura, función y composición de las moléculas biológicamente importantes.[1]
Biología molecular y otras ciencias
[editar]Esta área está relacionada con otros campos de la biología y la química, particularmente ingeniería genética y bioquímica. La biología molecular concierne principalmente al entendimiento de las interacciones de los diferentes sistemas de la célula, lo que incluye relaciones tales como las que existen entre el ADN y el ARN, la síntesis de proteínas, el metabolismo, y el cómo todas esas interacciones son reguladas para conseguir un correcto funcionamiento de la célula.[cita requerida]
La diferencia entre la química orgánica y la biología molecular o química biológica es que en la química biológica las moléculas de ADN tienen una historia y, por ende, en su estructura nos hablan de su historia, del pasado en el que se han constituido, mientras que una molécula orgánica, creada hoy, es solo testigo de su presente, sin pasado y sin evolución histórica.[2][3]
Características
[editar]
Al estudiar el comportamiento biológico de las moléculas que componen las células vivas, la Biología molecular roza otras ciencias que abordan temas similares: así, por ejemplo, juntamente con la Genética se interesa por la estructura y funcionamiento de los genes y por la regulación (inducción y represión) de la síntesis intracelular de enzimas y de otras proteínas. Con la Citología, se ocupa de la estructura de los corpúsculos subcelulares (núcleo, nucléolo, mitocondrias, ribosomas, lisosomas, etc.) y sus funciones dentro de la célula. Con la Bioquímica estudia la composición y cinética de las enzimas, interesándose por los tipos de catálisis enzimática, activaciones, inhibiciones competitivas o alostéricas, etc. También colabora con la Filogenética al estudiar la composición detallada de determinadas moléculas en las distintas especies de seres vivos, aportando valiosos datos para el conocimiento de la evolución.Sin embargo, difiere de todas estas ciencias enumeradas tanto en los objetivos concretos como en los métodos utilizados para lograrlos. Así como la Bioquímica investiga detalladamente los ciclos metabólicos y la integración y desintegración de las moléculas que componen los seres vivos, la Biología molecular pretende fijarse con preferencia en el comportamiento biológico de las macromoléculas (ADN, ARN, enzimas, hormonas, etc.) dentro de la célula y explicar las funciones biológicas del ser vivo por estas propiedades a nivel molecular.[cita requerida]
Métodos
[editar]Los métodos que emplea esta nueva ciencia son fundamentalmente los mismos que la Biofísica, Bioquímica, y Biología. Utiliza los análisis químicos, cualitativo y cuantitativo, los conocimientos de la Química orgánica, la Biología de microorganismos y de virus, etc., pero revisten especial importancia los nuevos microanalíticos tanto físicos como químicos. Merecen destacarse la microscopía electrónica, que permite resoluciones que alcanzan los 10 Amstrongs; la difracción de rayos X, que determina la estructura y disposición espacial de los átomos de las macromoléculas; la ultracentrifugación diferencial, tanto analítica como preparativa, que permite separaciones antes imposibles; la cromatografía de gases, y, en fase líquida, la espectrografía de infrarrojos, la Química con isótopos trazadores, la espectrometría de masas, etc.[cita requerida]
Técnicas
[editar]Clonación molecular
[editar]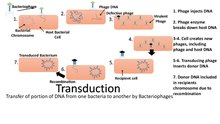
La clonación molecular se utiliza para aislar y luego transferir una secuencia de ADN de interés a un vector plásmido.[4] Esta tecnología de ADN recombinante se desarrolló por primera vez en la década de 1960.[5] En esta técnica, una secuencia de ADN que codifica una proteína de interés se clona mediante reacción en cadena de la polimerasa (PCR), y/o enzima de restricción, en un plásmido (vector de expresión). El plásmido vector suele tener al menos 3 características distintivas: un origen de replicación, un Sitio de Clonación Múltiple (SCM) y un marcador selectivo (normalmente resistencia a antibióticos). Además, aguas arriba del SCM se encuentran la región promotora y la transcripción, que regulan la expresión del gen clonado.
Este plásmido puede insertarse en células bacterianas o animales. La introducción de ADN en células bacterianas puede realizarse mediante transformación por captación de ADN desnudo, conjugación por contacto célula-célula o mediante transducción por vector viral. La introducción de ADN en células eucariotas, como las células animales, por medios físicos o químicos se denomina transfección. Existen diversas técnicas de transfección, como la transfección con fosfato cálcico, la electroporación, la microinyección y la transfección con liposomas. El plásmido puede integrarse en el genoma, dando lugar a una transfección estable, o puede permanecer independiente del genoma y expresarse temporalmente, lo que se denomina transfección transitoria.[6][7]
El ADN que codifica una proteína de interés se encuentra ahora dentro de una célula y la proteína puede expresarse. Existen diversos sistemas, como promotores inducibles y factores específicos de señalización celular, que ayudan a expresar la proteína de interés a niveles elevados. A continuación, se pueden extraer grandes cantidades de una proteína de la célula bacteriana o eucariota. La proteína puede someterse a pruebas de actividad enzimática en diversas situaciones, puede cristalizarse para estudiar su estructura terciaria o, en la industria farmacéutica, puede estudiarse la actividad de nuevos fármacos contra la proteína.[8]
Reacción en cadena de la polimerasa
[editar]La reacción en cadena de la polimerasa (PCR) es una técnica extremadamente versátil para copiar ADN. En pocas palabras, la PCR permite copiar o modificar una secuencia de ADN específica de formas predeterminadas. La reacción es extremadamente potente y, en condiciones perfectas, podría amplificar una molécula de ADN hasta convertirla en 1.070 millones de moléculas en menos de dos horas. La PCR tiene muchas aplicaciones, como el estudio de la expresión génica, la detección de microorganismos patógenos, la detección de mutaciones genéticas y la introducción de mutaciones en el ADN.[9] La técnica PCR puede utilizarse para introducir sitios de enzimas de restricción en los extremos de las moléculas de ADN, o para mutar bases particulares del ADN, este último es un método conocido como mutagénesis dirigida al sitio. La PCR también puede utilizarse para determinar si un fragmento concreto de ADN se encuentra en una biblioteca de ADNc. La PCR tiene muchas variaciones, como la PCR de transcripción inversa (RT-PCR) para la amplificación de ARN y, más recientemente, la PCR cuantitativa que permite la medición cuantitativa de moléculas de ADN o ARN.[10][11]

Electroforesis en gel
[editar]
La electroforesis en gel es una técnica que separa moléculas por su tamaño utilizando un gel de agarosa o poliacrilamida.[12] Esta técnica es una de las principales herramientas de la biología molecular. El principio básico es que los fragmentos de ADN se pueden separar aplicando una corriente eléctrica a través del gel - debido a que la columna vertebral del ADN contiene grupos fosfato cargados negativamente, el ADN migrará a través del gel de agarosa hacia el extremo positivo de la corriente.[12] Las proteínas también pueden separarse en función del tamaño utilizando un gel SDS-PAGE, o en función del tamaño y su carga eléctrica utilizando lo que se conoce como Electroforesis en gel 2D.[13].

Contenido
[editar]Al profundizar en cualquier fenómeno biológico y pretender explicar la naturaleza íntima de los procesos que determinan una propiedad o una función de los seres vivos, entramos inevitablemente en el campo de la Biología molecular. Veamos, por ejemplo el estudio de los genes. Las clásicas leyes de Mendel tienen su explicación inmediata en el conocimiento morfológico y funcional de los cromosomas. Pero cuando deseamos saber la composición y forma de actuación de un gen necesitamos penetrar a fondo en la estructura del ADN doble helicoide de Watson y Crick, el ordenamiento de bases púricas y pirimidímicas, es decir, la información genética.[14]

Al matizar la posibilidad de sintetizar una enzima por parte de un gen, debemos seguir el proceso de transmisión de esta información genética del ADN nuclear al ARN mensajero; la activación de los aminoácidos por el ARN transportador, la ordenación de estos aminoácidos activados sobre el ribosoma de acuerdo con la pauta prefijada por el ARN mensajero, la obtención de la estructura primaria de la enzima proteína. Todos estos temas son objeto de estudio de la Biología molecular. Pero hay más; la proteína, una vez sintetizada, debe ordenarse en el espacio según determinadas reglas que constituyen la conformación espacial específica (estructuras secundaria y terciaria) y a veces asociarse varias moléculas iguales o diferentes para constituir lo que se ha llamado estructuras cuaternaria y quinaria, de modo que las propiedades biológicas de la molécula como enzima están vinculadas a esta ordenación espacial compleja. La molécula proteica así organizada puede resultar ser una enzima que, en su actividad catalítica, es susceptible de sufrir activaciones o inhibiciones por determinadas sustancias, acciones éstas de trascendental importancia para la vida de la célula. Del mismo modo, la Biología molecular se interesa por la estructura química de las sustancias que componen las membranas biológicas y la ordenación de las enzimas que realizan acciones encadenadas, p. ej., dentro de las mitocondrias, núcleo y otros corpúsculos subcelulares, para explicar la mecánica de los ciclos y procesos bioquímicos determinados por la Topoquímica celular.[cita requerida]

Los procesos de reproducción de los virus, de las bacterias, y de los organismos superiores encierran multitud de incógnitas que trata de ir resolviendo la Biología molecular. Las mutaciones producidas por agentes físicos (rayos X, rayos gamma, calor, etc.) o químicos (sustancias mutágenas) tienen una explicación tanto más satisfactoria cuanto mejor se conoce la base molecular de los procesos de alteración en la estructura y ordenación de las bases nitrogenadas del ADN.[cita requerida]
El parentesco entre especies diferentes de seres vivos puede establecerse mediante el estudio individual comparado de las sustancias macromoleculares (proteínas) elaboradas por ellos. Así, de la secuencia de aminoácidos en la hemoglobina, mioglobina, citocromos, hormonas hipofisarias o insulina se induce el grado de proximidad filogenética, al demostrarse la evolución de la proteína por mutaciones progresivas. Multitud de fenómenos genéticos como selección natural, adaptación al ambiente, diferenciación de las especies, etc., tienen su última explicación a nivel molecular. Por último, la Biología molecular de microorganismos está aportando datos interesantes para la búsqueda de nuevos antibióticos y antimetabolitos, que permiten atacar eficaz y selectivamente a los gérmenes patógenos.[cita requerida]
Con todo esto no queremos afirmar que la Biología molecular sea una ciencia completa ni perfectamente elaborada. Todo lo contrario; los nuevos descubrimientos, al resolver una incógnita plantean muchos más interrogantes que son objeto de investigaciones futuras. Hoy día esta joven ciencia está en expansión explosiva. Por otro lado, la última y definitiva explicación de los comportamientos de las moléculas de los seres vivos requiere, para ser conocida en profundidad, enfrentarse con otras ramas de la ciencia tales como la Biofísica submolecular (orbitales, fuerzas de enlace, hibridación, etc.) e incluso la Física subatómica, para la cual se requiere un bagaje de conocimientos que jamás puede ser patrimonio de investigadores aislados, sino de equipos de trabajo científicamente heterogéneos, pero armónicamente conjuntados.[cita requerida]
Biólogos moleculares notables
[editar]- Francis Crick
- James Dewey Watson
- Rosalind Franklin
- Max Perutz
- François Jacob
- Christiane Nüsslein-Volhard
- Severo Ochoa
- Alberto Kornblihtt
- Linus Pauling
Referencias
[editar]- ↑ «Genome Glossary». Consultado el 30 de noviembre de 2012.
- ↑ Ilya Prigogine (2012). El nacimiento del tiempo. Buenos Aires, Fábula Tusquets editores. ISBN 978-987-670-087-0.
- ↑ Selya, Rena (17 de marzo de 2005). «Designs for Life: Molecular Biology after World War II (review)». Journal of Interdisciplinary History (en inglés) 35 (4): 646-647. ISSN 1530-9169. Consultado el 19 de octubre de 2018.
- ↑ «Fundamentos de la clonación molecular - Pasado, presente y futuro | NEB». www.neb.com. Consultado el 25 de noviembre de 2021.
- ↑ «Fundamentos de la clonación molecular - Pasado, presente y futuro | NEB». www.neb.com. Consultado el 4 de noviembre de 2021.
- ↑ Alberts, Bruce; Johnson, Alexander; Lewis, Julian; Raff, Martin; Roberts, Keith; Walter, Peter. Aislamiento, Clonación y secuenciación del ADN: //www. ncbi.nlm.nih.gov/books/NBK26837/.
- ↑ Lessard, Juliane C. (1 de enero de 2013). Molecular cloning. «Métodos de laboratorio en enzimología: DNA». Methods in Enzymology 529. pp. 85-98. ISBN 978-0-12-418687-3. ISSN 1557-7988. PMID 24011038. doi:10.1016/B978-0-12-418687-3.00007-0.
- ↑ Kokate, Chandrakant; Jalalpure, Sunil S.; Hurakadle, Pramod J. (2016). Libro de texto de Biotecnología Farmacéutica. Clonación de expresiones. Elsevier. p. 125. ISBN 9788131239872. Consultado el 8 de julio de 2019.
- ↑ Lenstra, J. A. (Julio de 1995). «Las aplicaciones de la reacción en cadena de la polimerasa en las ciencias de la vida». Cellular and Molecular Biology (Noisy-Le-Grand, Francia) 41 (5): 603-614. ISSN 0145-5680. PMID 7580841.
- ↑ «Reacción en cadena de la polimerasa (PCR)». National Center for Biotechnology Information. U.S. National Library of Medicine. Consultado el 31 de diciembre de 2016.
- ↑ «Hoja informativa sobre la reacción en cadena de la polimerasa (PCR)». Instituto Nacional de Investigación del Genoma Humano (NHGRI). Consultado el 31 de diciembre de 2016.
- ↑ a b Lee, Pei Yun; Costumbrado, John; Hsu, Chih-Yuan; Kim, Yong Hoon (2012- 04-20). «Electroforesis en gel de agarosa para la separación de fragmentos de ADN». Journal of Visualized Experiments (62): 3923. ISSN 1940-087X. PMC 4846332. PMID 22546956. doi:10.3791/3923.
- ↑ «Electroforesis en gel de agarosa para la separación de fragmentos de ADN». Journal of Visualized Experiments (62). April 2012. PMC 4846332. PMID 22546956. doi:10.3791/3923. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ 1928-, Watson, James D. (2011). La doble hélice relato personal del descubrimiento de la estructura del ADN (3a. ed edición). Alianza Editorial. ISBN 9788420674322. OCLC 776477600. Consultado el 19 de octubre de 2018.
- El contenido de este artículo incorpora material de la Gran Enciclopedia Rialp que mediante una autorización permitió agregar contenidos y publicarlos bajo licencia GFDL. La autorización fue revocada en abril de 2008, así que no se debe añadir más contenido de esta enciclopedia.
Bibliografía
[editar]- C. PETIT y G. PRÉVOST, Genetique et évolution, «Journal of Molecular Biologyu, Londres 1967; C. A. VILLE, Biología, México 1966.
- Genome Glossary. Información sobre proyecto de genoma humano (enlace roto disponible en Internet Archive; véase el historial, la primera versión y la última). (en inglés) (accedido el 15 de junio de 2009)
Enlaces externos
[editar] Wikiversidad alberga proyectos de aprendizaje sobre Biología molecular.
Wikiversidad alberga proyectos de aprendizaje sobre Biología molecular.
