Транспозоны
Транспозоны (англ. transposable element, transposon) — это участки ДНК организмов, способные к передвижению (транспозиции) и размножению в пределах генома[1]. Транспозоны также известны под названием «прыгающие гены» и являются примерами мобильных генетических элементов.
Транспозоны формально относятся к так называемой некодирующей части генома — той, которая в последовательности пар оснований ДНК не несёт информацию об аминокислотных последовательностях белков, хотя некоторые классы мобильных элементов содержат в своей последовательности информацию о ферментах, транскрибируются и катализируют передвижения; например, ДНК-транспозоны и ДДП-1 кодируют белки транспозаза, БОРС1 и БОРС2. У разных видов транспозоны распространены в разной степени: так, у человека транспозоны составляют до 45 % всей последовательности ДНК, у плодовой мухи Drosophila melanogaster часть мобильных элементов составляет лишь 15—20 % всего генома[2]. У растений транспозоны могут занимать основную часть генома — так, у кукурузы (Zea mays) с размером генома в 2,3 миллиардов пар оснований по крайней мере 85 % составляют различные мобильные элементы[3].
История открытия
Барбара Макклинток исследовала вариации окраски зерна и листьев кукурузы, и в 1948 году путём цитологических и генетических исследований пришла к выводу, что мобильные участки ДНК, Ac/Ds элементы, приводят к соматическому мозаицизму растений[4]. Она была первой, кто доказал, что геном эукариот не статичен, а содержит участки, которые могут передвигаться. В 1983 году за эту работу Барбара Макклинток получила Нобелевскую премию[5].
Хотя транспозоны были открыты в 1940-х годах, только через полвека стало понятно, насколько масштабным является их вклад в геном организмов. Так, получение первой нуклеотидной последовательности (секвенирование) генома человека показало, что мобильных элементов в последовательности ДНК не менее 50 %. Точную оценку получить трудно, поскольку некоторые транспозонные участки со временем настолько изменились, что их нельзя уверенно идентифицировать[6].
Поскольку транспозоны потенциально способны вызывать вредные мутации и поломки хроматина, с начала открытия мобильных элементов считалось, что их действие сводится к геномно-паразитической. Но в начале XXI столетия появляется всё больше данных о возможных благоприятных эффектах транспозонов для организмов[7], об эволюционном влиянии ретротранспозонов на геном плацентарных млекопитающих[8]. Идентифицируют случаи использования транспозонов организмами. Например, РНК ретротранспозона ДДП-1 участвует в образовании гетерохроматина во время инактивации X-хромосомы[9]. Плодовая муха не имеет теломеразы, а вместо этого использует обратную транскриптазу ретротранспозонов для продления теломерных участков, которые у Drosophila melanogaster представлены повторами транспозонов[10][11].
Типы транспозонов и механизмы их передвижения

Мобильные генетические элементы относятся к повторяющимся элементам генома — тем, которые имеют несколько копий в последовательности ДНК клетки. Повторяющиеся элементы генома могут располагаться в тандеме (микросателлиты, теломеры и т. д.) и могут быть рассеяны по геному (мобильные элементы, псевдогены и т. д.)[12].
Мобильные генетические элементы по типу транспозиции можно разделить на два класса: ДНК-транспозоны, которые применяют метод «вырезать и вставить», и ретротранспозоны, передвижение которых имеет в своем алгоритме синтез РНК из ДНК с последующим обратным синтезом ДНК из молекулы РНК, то есть метод «копировать и вставить».
Транспозоны также можно разделить по степени автономности. Как ДНК-транспозоны, так и ретротранспозоны имеют автономные и неавтономные элементы. Неавтономные элементы для транспозиции нуждаются в ферментах, которые кодируются автономными элементами, которые часто содержат значительно изменённые участки транспозонов и дополнительные последовательности. Количество неавтономных транспозонов в геноме может значительно превышать количество автономных[13].
ДНК-транспозоны

I. ДНК-транспозоны: способ передвижения «вырезать и вставить».
II. ДДП-1 ретротранспозоны: способ передвижения «копировать и вставить».
ДНК-транспозоны передвигаются по геному способом «вырезать и вставить» благодаря комплексу ферментов под названием транспозаза[1]. Информация об аминокислотной последовательности белка транспозазы закодирована в последовательности транспозона. Кроме того, этот участок ДНК может содержать другие, связанные с транспозоном последовательности, например гены или их части. Большинство ДНК-транспозонов имеют неполную последовательность. Такие транспозоны не являются автономными и передвигаются по геному благодаря транспозазе, которая закодирована другим, полным, ДНК-транспозоном[1].
На концах участков ДНК-транспозона расположены инвертированные повторы, которые являются особыми участками узнавания транспозазы, таким образом отличая эту часть генома от остальных. Транспозаза способна делать двухцепочные разрезы ДНК, вырезать и вставлять в ДНК-мишень транспозон[14].
К ДНК-транспозонам принадлежат Ac/Ds-элементы растений, которые были впервые открыты Барбарой Макклинток в кукурузе. Ac-элемент (англ. Activator) является автономным и кодирует транспозазу. Есть несколько типов Ds-элементов, которые способны к формированию разрывов хромосом и которые перемещаются по геному благодаря Ac-элементам[15].
Гелитроны — (англ. Helitron) тип транспозонов, который есть у растений, животных и грибов, но который широко представлен в геноме кукурузы, где он, в отличие от других организмов, находится в частях ДНК, богатых генами[3]. Гелитроны транспозируются по механизму «катящегося кольца» (англ. rolling circle). Процесс начинается с разрыва одной цепи ДНК-транспозоны. Высвобожденный участок ДНК вторгается в последовательность-мишень, где формируется гетеродуплекс. С помощью ДНК-репликации завершается внедрение транспозона в новый участок[16].
Гелитроны могут захватывать соседние последовательности при транспозиции.
Ретротранспозоны
Ретротранспозоны — это мобильные генетические элементы, которые применяют метод «копировать и вставить» для распространения в геноме животных[17]. По крайней мере 45 % генома человека составляют ретротранспозоны и их производные. Процесс передвижения включает промежуточную стадию молекулы РНК, которая считывается с участка ретротранспозона и которая затем, в свою очередь, используется как матрица для обратной транскрипции в последовательность ДНК. Новосинтезированный ретротранспозон встраивается в другой участок генома.
Активные ретротранспозоны млекопитающих делятся на три основные семьи: Alu-повторы, ДДП-1, SVA.
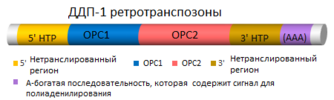
- ДДП-1 ретротранспозоны — длинные диспергированные повторы — тип ретротранспозонов, который широко распространён у млекопитающих и составляет до 20 % генома. ДДП-1 элементы имеют длину около 6 тысяч пар оснований[7]. Большинство этих ретротранспозонов в геноме представлено неполно, хотя существует примерно 150 полных и потенциально мобильных ДДП-1 элементов в последовательности ДНК человека и примерно 3000 — у мыши[7].
Процесс передвижения начинается со считывания молекулы РНК с элемента ДДП-1. РНК транспортируется к цитоплазме, где от неё транслируются белки БОРС1 (который является РНК-связывающим белком) и БОРС2 (который является белком с эндонуклеазной и возвратно-транскриптазной активностями). БОРС1, БОРС2 и РНК транспозона формируют рибонуклеопротеин и импортируются в ядро, где происходит обратная транскрипция ретротранспозона[18].
Большинство случаев вставки ДДП-1 элементов происходит не до конца, и такие копии больше не способны к самостоятельной мобилизации[7].
Существуют сведения о неканонических функциях ДДП-1 элементов во время инактивации X-хромосомы[9]. - ДКП — длинные концевые повторы — ретротранспозоны, имеющие конечные повторяющиеся последовательности, которые играют важную роль в транскрипции и обратной транскрипции РНК транспозона[4]. ДКП-элементы кодируют белки pol и gag, которые близки к белкам ретровирусов, но, в отличие от последних, ДКП не хватает белков, которые смогли бы сформировать внешнюю оболочку (суперкапсид) и выйти из клетки[13].
- КДП — короткие диспергированные повторы являются неавтономными ретротранспозонами: они требуют активности ДДП-1 элементов для передвижения, в ДНК-последовательности КДП содержат только участок связывания РНК-полимеразы[4]. В число КДП входят Alu-ретротранспозоны.

- Alu-повтор (Alu от Arthrobacter luteus) — широко распространённые мобильные элементы в геноме человека[19]. Alu-элементы имеют длину около 300 пар оснований и часто расположены в интронах, участках генома, которые не транслируются, и межгенных участках[12]. Приставку Alu- ретротранспозоны получили за то, что они содержат последовательность распознавания рестрикционного энзима AluI[12]. Анализ последовательностей показал, что Alu-элементы возникли у приматов примерно 65 миллионов лет назад от гена 7SL РНК, который входит в рибосомный комплекс[12]. Alu-ретротранспозоны не имеют собственной обратной транскриптазы, поэтому для передвижения им необходимые ферменты ДДП-1 элементов.
Alu-элементы являются участками, где происходит до 90 % всех случаев A-I редактирования РНК[18]. - SVA — мобильные элементы длиной в 2-3 тысячи пар оснований ДНК, состоящие из нескольких частей: коротких разбросанных элементов (КДП), вариабельного числа тандемных повторов (ВЧТП), Alu-последовательностиі[20] и CT-повтора, с последовательностью CCCTCT, которая встречается чаще всего и имеет название гексамер (Hex)[21]. SVA элементы значительно варьируют в длину из-за разного количества составляющих повторов[21]. Они не являются автономными и нуждаются в белках, закодированных в ДДП1 ретротранспозонах для передвижения, но они активны в геноме человека[4]. SVA-элементы претерпевают высокий уровень метилирования ДНК в большинстве тканей человека. Интересным фактом является заниженное метилирование ДНК SVA-ретротранспозонов в мужских половых клетках человека, тогда как у шимпанзе SVA-последовательности сперматозоидов высоко метилированы[22].
Механизмы блокировки транспозонов
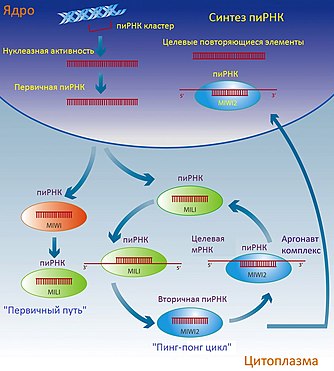
Мобильные элементы генома достаточно широко представлены в растительных и животных геномах. Их высокая активность является риском для стабильности генома, поэтому их экспрессия жестко регулируется, особенно в тех тканях, которые принимают участие в формировании гамет и передаче наследственной информации потомкам. У растений и животных регуляция активности мобильных элементов генома происходит путём de novo метилирования последовательности ДНК и активности некодирующих РНК вместе с белковыми комплексами Аргонавт[23].
Основная роль малых некодирующих РНК, которые взаимодействуют с пиви-комплексом, или пиРНК, заключается в подавлении мобильных элементов генома в зародышевых тканях. Эта роль пиРНК достаточно высоко консервативна у животных[24].
У мышей мобильные элементы генома на протяжении онтогенеза находятся преимущественно в неактивном состоянии, которое достигается путём эпигенетических взаимодействий и активности некодирующих РНК[25]. В период эмбрионального развития эпигенетическая метка метилирования ДНК подвергается репрограммированию: родительские метки стираются, а новые устанавливаются[26]. В этот период часть белков-аргонавтов — пиви-белки (Mili и Miwi2) — и некодирующие РНК, которые с ними взаимодействуют — пиРНК — играют ключевую роль в de novo подавлении ретротранспозонов мышей путём метилирования ДНК, и пинг-понг цикла пиРНК амплификации, и подавления мишени[27]. Если у мышей возникает недостаток белков Mili и Miwi2, это приводит к активации ДДП-1 и ДКП и остановке гаметогенеза и стерильности у самцов[24]. Недавние работы показали, что у мухи Drosophila melanogaster активным кофактором в подавлении является белок СФГ-1.
Механизм пиРНК-индуцированного подавления транспозонов окончательно не выяснен, но схематически его можно представить такой моделью[28]:
- первичное накопление одноцепочечных молекул РНК, пиРНК-прекурсоров;
- созревание пиРНК и их амплификация с помощью пиви-белков (пинг-понг цикл);
- подавление целевого транспозона, что может происходить несколькими путями: деградация РНК (с помощью РНКазной активности H-подобного домена белков-аргонавтов), подавление трансляции и привлечение хроматин-модифицирующих систем (таких, как SWI/SNF белки[13]) и дальнейшее эпигенетическое подавление транспозона.
В отличие от вирусов, которые используют организм хозяина для размножения и способны его покинуть, мобильные генетические элементы существуют исключительно в организме хозяина. До некоторой степени поэтому транспозоны способны регулировать свою активность. Примером этого является Ac ДНК-транспозоны — автономные мобильные элементы растений, кодирующие собственную транспозазу. Ac-элементы проявляют способность снижать активность транспозазы при увеличении её копий[29].
Также подавление растительных автономных ДНК-транспозонов MuDR может происходить с помощью Muk. Muk является вариантом MuDR и имеет в своей последовательности несколько палиндромных участков ДНК. Когда Muk транскрибируется, такая РНК формирует шпильку, затем режется комплексом ферментов на малые интерферирующие РНК (миРНК), которые заглушают активность MuDR с помощью процесса РНК-интерференции[29].
Болезни
По состоянию на 2012 год задокументировано 96 различных заболеваний человека, причиной которых является de novo внедрение мобильных генетических элементов[22]. Alu-повторы часто вызывают хромосомные аберрации и являются причиной 50 разновидностей заболеваний[30]. Так, у нейрофиброматоза I типа было найдено 18 случаев встроенных ретротранспозонов, 6 из которых происходят в 3 специфических местах. Активность ДДП-1 мобильных элементов в соматических тканях зафиксирована у пациентов с раком легких[22].
Если транспозиция, которая вызывает заболевания, происходит в гаметах, то следующие поколения наследуют болезни. Так, гемофилия может возникать из-за встраивания ретротранспозона ДДП-1 в участок ДНК, кодирующий ген VIII фактора свертывания крови. У мышей были зафиксированы случаи онкогенеза, остановки развития и стерильность в связи со встраиванием мобильных элементов генома[30].
Эволюционная роль транспозонов
Некоторые этапы эволюционирования организмов были вызваны активностью мобильных элементов генома. Уже первая нуклеотидная последовательность генома человека доказала, что многие гены были производными транспозонов[6]. Мобильные элементы генома могут влиять на организацию генома путём рекомбинации генетических последовательностей и входя в состав таких фундаментальных структурных элементов хроматина, как центромеры и теломеры[31]. Мобильные элементы могут влиять на соседние гены, меняя узоры (паттерны) сплайсинга и полиаденилирования или выполняя функции энхансеров или промоторов[13]. Транспозоны могут влиять на структуру и функции генов путём выключения и изменения функций, изменения структуры генов, мобилизации и реорганизации фрагментов генов и изменения эпигенетического контроля генов[16].
Репликация транспозонов может вызвать некоторые заболевания, но, несмотря на это, в процессе эволюции транспозоны не были удалены и остались в ДНК-последовательностях почти всех организмов, или в виде целых копий, которые имели возможность передвигаться по ДНК, или в укороченном виде, потеряв способность к передвижению. Но укороченные копии также могут принимать участие в таких процессах, как пост-транскрипционная регуляция генов, рекомбинация и т. п.[31] Также важным моментом в потенциальной способности транспозонов влиять на темпы эволюции является то, что их регуляция зависит от эпигенетических факторов. Это приводит к возможности транспозонов реагировать на изменения окружающей среды и вызывать генетическую нестабильность[31]. На стресс транспозоны активируются или прямо, или путём снижения их подавления белками-аргонавтами и пиРНК[13]. У растений мобильные генетические элементы очень чувствительны к различным типам стресса, на их активность могут влиять многочисленные абиотические и биотические факторы, среди которых солёность, ранения, холод, тепло, бактериальные и вирусные инфекции[16].
Ещё одним возможным механизмом эволюции геномов организмов является горизонтальный перенос генов — процесс передачи генов между организмами, которые не находятся в отношениях «предки-потомки». Есть сведения о том, что взаимодействия паразитических организмов и животных-хозяев могут повлечь горизонтальный перенос генов с помощью транспозонов, который состоялся между позвоночными и беспозвоночными организмами[32].
Примеры эволюционной роли мобильных генетических элементов
Считается, что приобретённый иммунитет млекопитающих возник у челюстных рыб примерно 500 миллионов лет назад[33]. Приобретённый иммунитет позволяет формировать антитела для многих видов патогенов, попадающих в организм млекопитающих, включая человека. Для формирования различных антител клетки иммунной системы изменяют последовательность ДНК путём соматической рекомбинации с помощью системы, которая возникла и эволюционировала благодаря мобильным элементам генома[33].
Нейроны, клетки нервной системы, могут иметь мозаичный геном, то есть последовательность ДНК у них отличается от последовательности ДНК других клеток, хотя все они сформировались из одной клетки-предшественника — зиготы. Доказано, что у крыс специально вставленные ДДП-1 ретротранспозоны человека активны даже в зрелом возрасте. Также зафиксировано увеличение копий ДДП-1 ретротранспозонов в нейронах некоторых участков мозга, в частности гипоталамуса, по сравнению с другими тканями у взрослых людей[34]. Также установлено, что мобильные элементы приводят к разнородности в нейронах мухи Drosophila melanogaster[2]. Активность мобильных элементов в нейронах может повлечь синаптическую пластичность и большую вариабельность поведенческих реакций[7].
Последовательности ДНК генов теломеразы и ДДП-1 ретротранспозонов имеют высокую гомологию, что свидетельствует о возможности происхождения теломераз от ретротранспозонов[1].
У растений очень большая скорость эволюции геномов, поэтому лучше всего известны те влияния мобильных элементов, которые возникли вследствие одомашнивания, поскольку оно произошло недавно, и эти изменения легко идентифицировать, поскольку известны черты, по которым велась селекция культурных растений[16]. Примером может быть приобретение овальной формы римским помидором Solanum lycopersicum. Ген, который находится в локусе SUN, был перемещён путём ретротранспозиции в другой участок ДНК, где он регулируется другими промоторными последовательностями у овальных томатов[16].
Использование транспозонов
Генная инженерия
Поскольку мобильные элементы генома способны к встраиванию в хроматин, они используются в генной инженерии для специального и контролируемого встраивания генов или участков ДНК, которые изучают учёные. Транспозоны используются для мутагенеза и для определения регуляторных элементов генома в лабораториях.
Наиболее известная система для ввёденного мутагенеза in vivo — P-мобильный элемент мухи D. melanogaster, с помощью которого можно изучать функции генов, налаживание хромосомных аберраций и т. п.[35]
У позвоночных животных долгое время не было эффективной методики транспозонной модификации генома. Сейчас есть система Tol2 мобильного элемента, полученная из японской рыбы Oryzias latipes, которая используется как у мышей, так и на клеточных линиях человека[35]. Также успешной является система транспозонов Minos[36].
Система транспозонов Спящая Красавица (англ. Sleeping Beauty) была создана на основе последовательности ДНК транспозазы из рыбы. Удачное использовании этой системы на мышах позволило определить кандидатов в онкогены рака кишечника человека[37].
Филогенетика
Кроме использования транспозонов в генной инженерии, изучение активности транспозонов является методом филогенетики. Путём анализа и сопоставления нуклеотидных последовательностей геномов различных видов можно найти транспозоны, которые имеются у одних видов, но отсутствуют у других. Виды, у которых есть одинаковый ретротранспозон, скорее всего получили его от общего предка. Таким образом, можно получить информацию об эволюционном развитии видов и строить филогенетические деревья[38].
Примечания
- ↑ 1 2 3 4 Сиволоб А. В. Молекулярна біологія. — Київ: Видавничо-поліграфічний центр «Київський університет», 2008. — 384 с.
- ↑ 1 2 Perrat P. N., DasGupta S., Wang J.; et al. (2013). "Transposition-Driven Genomic Heterogeneity in the Drosophila Brain". Science. 340 (6128): 91—95. doi:10.1126/science.1231965. ISSN 0036-8075.
{{cite journal}}: Явное указание et al. в:|author=(справка)Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ 1 2 Patrick S. Schnable, Doreen Ware, Robert S. Fulton; et al. (2009). "The B73 maize genome: complexity, diversity, and dynamics". Science. 326 (5956): 1112—1115. doi:10.1126/science.1178534. PMID 19965430.
{{cite journal}}: Явное указание et al. в:|author=(справка)Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ 1 2 3 4 Levin Henry L., Moran John V. (2011). "Dynamic interactions between transposable elements and their hosts". Nature Reviews Genetics. 12 (9): 615—627. doi:10.1038/nrg3030. ISSN 1471-0056.
- ↑ "Nobel Prize to Barbara McClintock". Nature. 305 (5935): 575—575. 1983. doi:10.1038/305575a0. ISSN 0028-0836.
- ↑ 1 2 Lander Eric S., Linton Lauren M., Birren Bruce. (2001). "Initial sequencing and analysis of the human genome". Nature. 409 (6822): 860—921. doi:10.1038/35057062. ISSN 0028-0836.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ 1 2 3 4 5 Singer Tatjana, McConnell Michael J., Marchetto Maria C.N.; et al. (2010). "LINE-1 retrotransposons: mediators of somatic variation in neuronal genomes?". Trends in Neurosciences. 33 (8): 345—354. doi:10.1016/j.tins.2010.04.001. ISSN 0166-2236.
{{cite journal}}: Явное указание et al. в:|author=(справка)Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Kaneko-Ishino Tomoko, Ishino Fumitoshi. (2010). "Retrotransposon silencing by DNA methylation contributed to the evolution of placentation and genomic imprinting in mammals". Development, Growth & Differentiation. 52 (6): 533—543. doi:10.1111/j.1440-169X.2010.01194.x. ISSN 0012-1592.
- ↑ 1 2 Melamed Esther, Arnold Arthur P. (2009). "The role of LINEs and CpG islands in dosage compensation on the chicken Z chromosome". Chromosome Research. 17 (6): 727—736. doi:10.1007/s10577-009-9068-4. ISSN 0967-3849.
- ↑ Abad J. P. (2004). "TAHRE, a Novel Telomeric Retrotransposon from Drosophila melanogaster, Reveals the Origin of Drosophila Telomeres". Molecular Biology and Evolution. 21 (9): 1620—1624. doi:10.1093/molbev/msh180. ISSN 0737-4038.
- ↑ Nick Fulcher, Elisa Derboven, Sona Valuchova & Karel Riha. (2013). "If the cap fits, wear it: an overview of telomeric structures over evolution". Cellular and molecular life sciences : CMLS. doi:10.1007/s00018-013-1469-z. PMID 24042202.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ 1 2 3 4 Batzer Mark A., Deininger Prescott L. (2002). "Alu repeats and human genomic diversity". Nature Reviews Genetics. 3 (5): 370—379. doi:10.1038/nrg798. ISSN 1471-0056.
- ↑ 1 2 3 4 5 R. Keith Slotkin, Robert Martienssen. (2007). "Transposable elements and the epigenetic regulation of the genome". Nature reviews. Genetics. 8 (4): 272—285. doi:10.1038/nrg2072. PMID 17363976.
{{cite journal}}: Неизвестный параметр|month=игнорируется (справка) - ↑ van Opijnen Tim, Camilli Andrew. (2013). "Transposon insertion sequencing: a new tool for systems-level analysis of microorganisms". Nature Reviews Microbiology. 11 (7): 435—442. doi:10.1038/nrmicro3033. ISSN 1740-1526.
- ↑ Chunguang Du, Andrew Hoffman, Limei He, Jason Caronna & Hugo K. Dooner (2011). "The complete Ac/Ds transposon family of maize". BMC genomics. 12: 588. doi:10.1186/1471-2164-12-588. PMID 22132901.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) Википедия:Обслуживание CS1 (не помеченный открытым DOI) (ссылка) - ↑ 1 2 3 4 5 Damon Lisch (2013). "How important are transposons for plant evolution?". Nature reviews. Genetics. 14 (1): 49—61. doi:10.1038/nrg3374. PMID 23247435.
- ↑ Baillie J. Kenneth, Barnett Mark W., Upton Kyle R. (2011). "Somatic retrotransposition alters the genetic landscape of the human brain". Nature. 479 (7374): 534—537. doi:10.1038/nature10531. ISSN 0028-0836.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ 1 2 Cordaux Richard, Batzer Mark A. (2009). "The impact of retrotransposons on human genome evolution". Nature Reviews Genetics. 10 (10): 691—703. doi:10.1038/nrg2640. ISSN 1471-0056.
- ↑ Stower Hannah. (2013). "Alternative splicing: Regulating Alu element 'exonization'". Nature Reviews Genetics. 14 (3): 152—153. doi:10.1038/nrg3428. ISSN 1471-0056.
- ↑ Varki Ajit, Geschwind Daniel H., Eichler Evan E. (2008). "Human uniqueness: genome interactions with environment, behaviour and culture". Nature Reviews Genetics. 9 (10): 749—763. doi:10.1038/nrg2428. ISSN 1471-0056.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ 1 2 Hancks D. C., Mandal P. K., Cheung L. E.; et al. (2012). "The Minimal Active Human SVA Retrotransposon Requires Only the 5'-Hexamer and Alu-Like Domains". Molecular and Cellular Biology. 32 (22): 4718—4726. doi:10.1128/MCB.00860-12. ISSN 0270-7306.
{{cite journal}}: Явное указание et al. в:|author=(справка)Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ 1 2 3 Hancks Dustin C., Kazazian Haig H. (2012). "Active human retrotransposons: variation and disease". Current Opinion in Genetics & Development. 22 (3): 191—203. doi:10.1016/j.gde.2012.02.006. ISSN 0959-437X.
- ↑ Law Julie A., Jacobsen Steven E. (2010). "Establishing, maintaining and modifying DNA methylation patterns in plants and animals". Nature Reviews Genetics. 11 (3): 204—220. doi:10.1038/nrg2719. ISSN 1471-0056.
- ↑ 1 2 Siomi Mikiko C., Sato Kaoru, Pezic Dubravka; et al. (2011). "PIWI-interacting small RNAs: the vanguard of genome defence". Nature Reviews Molecular Cell Biology. 12 (4): 246—258. doi:10.1038/nrm3089. ISSN 1471-0072.
{{cite journal}}: Явное указание et al. в:|author=(справка)Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ De Fazio Serena, Bartonicek Nenad, Di Giacomo Monica. (2011). "The endonuclease activity of Mili fuels piRNA amplification that silences LINE1 elements". Nature. 480 (7376): 259—263. doi:10.1038/nature10547. ISSN 0028-0836.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Popp Christian, Dean Wendy, Feng Suhua. (2010). "Genome-wide erasure of DNA methylation in mouse primordial germ cells is affected by AID deficiency". Nature. 463 (7284): 1101—1105. doi:10.1038/nature08829. ISSN 0028-0836.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Castel Stephane E., Martienssen Robert A. (2013). "RNA interference in the nucleus: roles for small RNAs in transcription, epigenetics and beyond". Nature Reviews Genetics. 14 (2): 100—112. doi:10.1038/nrg3355. ISSN 1471-0056.
- ↑ Luteijn Maartje J., Ketting René F. (2013). "PIWI-interacting RNAs: from generation to transgenerational epigenetics". Nature Reviews Genetics. 14 (8): 523—534. doi:10.1038/nrg3495. ISSN 1471-0056.
- ↑ 1 2 Damon Lisch (2012). "Regulation of transposable elements in maize". Current opinion in plant biology. 15 (5): 511—516. doi:10.1016/j.pbi.2012.07.001. PMID 22824142.
- ↑ 1 2 Zamudio N, Bourc'his D. (2010). "Transposable elements in the mammalian germline: a comfortable niche or a deadly trap?". Heredity. 105 (1): 92—104. doi:10.1038/hdy.2010.53. ISSN 0018-067X.
- ↑ 1 2 3 Rebollo Rita, Horard Béatrice, Hubert Benjamin; et al. (2010). "Jumping genes and epigenetics: Towards new species". Gene. 454 (1–2): 1—7. doi:10.1016/j.gene.2010.01.003. ISSN 0378-1119.
{{cite journal}}: Явное указание et al. в:|author=(справка)Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Gilbert Clément, Schaack Sarah, Pace II John K.; et al. (2010). "A role for host–parasite interactions in the horizontal transfer of transposons across phyla". Nature. 464 (7293): 1347—1350. doi:10.1038/nature08939. ISSN 0028-0836.
{{cite journal}}: Явное указание et al. в:|author=(справка)Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ 1 2 Flajnik Martin F., Kasahara Masanori. (2009). "Origin and evolution of the adaptive immune system: genetic events and selective pressures". Nature Reviews Genetics. 11 (1): 47—59. doi:10.1038/nrg2703. ISSN 1471-0056.
- ↑ Coufal Nicole G., Garcia-Perez José L., Peng Grace E. (2009). "L1 retrotransposition in human neural progenitor cells". Nature. 460 (7259): 1127—1131. doi:10.1038/nature08248. ISSN 0028-0836.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ 1 2 Carlson Corey M., Largaespada David A. (2005). "Insertional mutagenesis in mice: new perspectives and tools". Nature Reviews Genetics. 6 (7): 568—580. doi:10.1038/nrg1638. ISSN 1471-0056.
- ↑ Venken Koen J T, Schulze Karen L, Haelterman Nele A. (2011). "MiMIC: a highly versatile transposon insertion resource for engineering Drosophila melanogaster genes". Nature Methods. 8 (9): 737—743. doi:10.1038/nmeth.1662. ISSN 1548-7091.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ March H Nikki, Rust Alistair G, Wright Nicholas A. (2011). "Insertional mutagenesis identifies multiple networks of cooperating genes driving intestinal tumorigenesis". Nature Genetics. 43 (12): 1202—1209. doi:10.1038/ng.990. ISSN 1061-4036.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Inferring Phylogenetic Trees from Transposon Data
Глоссарий
- Ac/Ds — система активации-диссоциации (англ. Activator/Dissociation system)
- БОРС1 — белок первой открытой рамки считывания (англ. Open reading frame 1 protein, ORF1p); БОРС2 — белок второй открытой рамки считывания
- ДДП — длинные диспергированные повторы (англ. Long INterspersed Elements, LINE, L)
- ДКП — длинные концевые повторы (англ. Long Terminal Repeat, LTR)
- КДП — короткие диспергированные повторы (англ. Short INterspersed Elements, SINE)
- SVA (англ. SINE-R-VNTR-Alu) — элементы КДП-ВЧТП-Alu (где ВЧТП — варьирующие по числу тандемные повторы, англ. Variable number of tandem repeat, VNTR).
- СФГ-1 — специфический фактор гаметоцитов-1 (англ. gametocyte-specific factor 1, GTSF1)
Литература
- Голубовский М. Д. Нестабильность генов и мобильные элементы: к истории изучения и открытия // Историко-биологические исследования. 2011. № 4. С.60-78.
- Титов И.И., Ворожейкин П.С. миРНК-содержащие транспозоны человека // Вавиловский журнал генетики и селекции. — Т. 15, № 323 — 326.
- Авдеев Ю. И., Авдеев А. Ю. Обнаружение транспозонов половых клеток у спонтанных мутантов solanum lycopersicum L. и селекция растений // нет. 2013. № 2. С.103-109.
- Данилевская О. Н. Мобильные генетические элементы дрозофилы: история открытия и судьба первооткрывателей // Историко-биологические исследования. 2011. № 4. С.79-89.
- Юрченко Н. Н., Коваленко Л. В., Захаров И. К. Мобильные генетические элементы: нестабильность генов и геномов // Вавиловский журнал генетики и селекции. — Т. 15, № 2. — С. 261 — 270.