基因家族:修订间差异
Luckas-bot(留言 | 贡献) 小 機器人 新增: cs:Genová rodina |
补救2个来源,并将0个来源标记为失效。) #IABot (v2.0.9.5 |
||
| (未显示9个用户的29个中间版本) | |||
| 第1行: | 第1行: | ||
[[File:Phylogenetic tree of Mups.jpg|thumbnail|Mup基因家族的系统发生树]] |
|||
'''基因家族'''(gene family),是来源于同一个祖先,由一个[[基因]]通过基因重复而产生两个或更多的拷贝而构成的一组基因,它们在结构和功能上具有明显的相似性,编码相似的[[蛋白质]]产物,<ref>朱玉贤等 《分子生物学》 高等教育出版社 ISBN 978-7-04-022214-2</ref> |
|||
'''基因家族'''({{lang-en|Gene family}}),是来源于同一个祖先,由一个[[基因]]通过基因重复而产生两个或更多的拷贝而构成的一组基因,它们在结构和功能上具有明显的相似性,编码相似的[[蛋白质]]产物。<ref>朱玉贤等 《分子生物学》 高等教育出版社 ISBN 978-7-04-022214-2</ref>一个这样的家族是人类[[血红蛋白]]亚基的基因,10个基因在不同染色体上的两个簇中,称为α-珠蛋白和β-珠蛋白基因座。这两个[[基因簇]]被认为是由于前体基因在大约5亿年前被复制而产生的<ref>{{Cite book|title = Thompson & Thompson Genetics in Medicine|last = Nussbaum|first = Robert L.|publisher = Elsevier|year = 2016|isbn = 978-1-4377-0696-3|location = Philadelphia, PA|pages = 25|last2 = McInnes|first2 = Roderick R.|last3 = Willard|first3 = Huntington F.|edition = 8th}}</ref>。 |
|||
[[基因]]根据共享的[[核苷酸]]或[[蛋白质序列]]被分为家族。 [[系統發生樹|系統發生]]技术可以用作更严格的测试。[[外显子]]在编码序列中的位置可用于推断共同的祖先。 了解基因编码的蛋白质序列可以让研究人员应用方法来发现[[蛋白质]]序列之间的相似性,这些相似性提供的信息比[[DNA]]序列之间的相似性或差异性更多。 |
|||
如果一个基因家族的基因编码蛋白质,那么术语'''[[蛋白质家族]]'''通常以与'''基因家族'''类似的方式使用。 |
|||
基因家族沿特定谱系的扩展或收缩可能是偶然的,也可能是自然选择的结果<ref>Hartl, D.L. and Clark A.G. 2007. Principles of population genetics. Chapter 7, page 372.</ref>。区分这两种情况在实践中往往很困难。 最近的工作使用统计模型和算法技术的组合来检测受自然选择影响的基因家族<ref>{{cite journal|last1=Demuth|first1=Jeffery P.|last2=Bie|first2=Tijl De|last3=Stajich|first3=Jason E.|last4=Cristianini|first4=Nello|last5=Hahn|first5=Matthew W.|last6=Borevitz|first6=Justin|title=The Evolution of Mammalian Gene Families|journal=PLOS ONE|date=20 December 2006|volume=1|issue=1|pages=e85|doi=10.1371/journal.pone.0000085|pmid=17183716|pmc=1762380|bibcode=2006PLoSO...1...85D|doi-access=free}}</ref>。 |
|||
[[国际人类基因组组织|国际人类基因组组织(HUGO)]]的{{le|人类基因组组织基因命名委员会|HUGO Gene Nomenclature Committee}}(HGNC) 使用基因家族成员的“主干”(或“根”)符号(通过[[同源]]性或功能)创建命名方案,并使用分层编号系统来区分各个成员<ref name=hgnc>{{cite journal|last1=Daugherty|first1=LC|last2=Seal|first2=RL|last3=Wright|first3=MW|last4=Bruford|first4=EA|title=Gene family matters: expanding the HGNC resource.|journal=Human Genomics|date=Jul 5, 2012|volume=6|issue=1|pages=4|pmid=23245209|doi=10.1186/1479-7364-6-4|pmc=3437568}}</ref><ref name="HGNC_gene_families_help">{{Cite web |author=HGNC |author-link=HUGO Gene Nomenclature Committee |title=Gene group help |url=https://www.genenames.org/help/genegroup |access-date=2020-10-13 |archive-date=2023-06-01 |archive-url=https://web.archive.org/web/20230601160259/https://www.genenames.org/help/genegroup/ |dead-url=no }}</ref>。 例如,对于[[过氧化物氧还酶]](Peroxiredoxin)家族,PRDX 是根符号,家族成员为 PRDX1、PRDX2、PRDX3、PRDX4、PRDX5 和 PRDX6。 |
|||
同一家族基因可以紧密排列在一起,形成一个[[基因簇]],但多数时候,它们是分散在同一[[染色体]]的不同位置,或者存在于不同的染色体上的,各自具有不同的表达调控模式。 |
同一家族基因可以紧密排列在一起,形成一个[[基因簇]],但多数时候,它们是分散在同一[[染色体]]的不同位置,或者存在于不同的染色体上的,各自具有不同的表达调控模式。 |
||
== |
== 基本结构 == |
||
[[File:Ortholog paralog analog examples.svg|thumb|400x400px|Gene [[Phylogenetic tree|phylogeny]] as lines within grey species phylogeny. Top: An ancestral [[gene duplication]] produces two paralogs ([[histone H1.1]] and [[Histone H1.2|1.2]]). A speciation event produces orthologs in the two daughter species (human and chimpanzee). Bottom: in a separate species ([[E. coli]]), an gene has a similar function ([[histone-like nucleoid-structuring protein]]) but has a separate evolutionary origin and so is an [[Analogy (biology)|analog]].]] |
|||
<references/> |
|||
基因组组织的一个层次是将基因分组到几个基因家族中<ref name=":1GenesToGenomes">{{cite book|first1=Leland H.|last1=Hartwell|display-authors=etal|title=Genetics : from genes to genomes|date=2011|publisher=McGraw-Hill|location=New York|isbn=978-0073525266|edition=4th}}</ref><ref name=":4LifeAndDeath">{{cite journal|last1=Demuth|first1=JP|last2=Hahn|first2=MW|title=The life and death of gene families.|journal=BioEssays|date=January 2009|volume=31|issue=1|pages=29–39|pmid=19153999|doi=10.1002/bies.080085|s2cid=9528185}}</ref>。 基因家族是共享共同祖先的相关基因组。 基因家族的成员可能是旁系同源物或直系同源物。 基因旁系同源物是同一物种中具有相似序列的基因,而基因直系同源物是不同物种中具有相似序列的基因。 基因家族在大小、序列多样性和排列方面变化很大。 根据家族内基因的多样性和功能,家族可分为多基因家族或超家族<ref name=":1GenesToGenomes" /><ref name=":2MultifamAndSuperfam">{{cite journal|last1=Ohta|first1=Tomoka|title=Gene families: multigene families and superfamilies|journal=eLS|date=2008|doi=10.1038/npg.els.0005126|isbn=978-0470015902}}</ref>。 |
|||
'''多基因'''家族通常由具有相似序列和功能的成员组成,尽管高度差异(在序列和/或功能水平上)不会导致基因从基因家族中移除。 家族中的单个基因可能在同一条染色体上紧密排列在一起,也可能分散在不同染色体上的整个基因组中。 由于它们序列的相似性和重叠功能,家族中的单个基因通常共享调控元件<ref name=":1GenesToGenomes" /><ref name=":2MultifamAndSuperfam" />。在某些情况下,基因成员具有相同(或几乎相同)的序列。 这些家族允许根据需要在短时间内表达大量基因产物。 其他家族允许在不同细胞类型或生物体发育的不同阶段表达相似但特定的产物<ref name=":1GenesToGenomes" />。 |
|||
'''超家族'''比单个多基因家族大得多。 超家族包含多达数百个基因,包括多个多基因家族以及单个基因成员。 大量的成员使得超家族可以广泛分散,一些基因聚集在一起,一些基因分散得很远。 这些基因在序列和功能上是多种多样的,显示出不同的表达水平和独立的调节控制<ref name=":1GenesToGenomes" /><ref name=":2MultifamAndSuperfam" />。 |
|||
== 演化 == |
|||
基因家族是基因组中信息存储层次结构的一部分,在多细胞生物的演化和多样性中发挥着重要作用。 基因家族是信息和遗传变异的大单位<ref name=":1GenesToGenomes" />。 随着演化时间的推移,基因家族随着新基因家族的形成和一些基因家族的丢失而扩大和收缩。在几个演化谱系中,基因以相对相同的速率获得和丢失。 当自然选择有利于额外的基因拷贝时,就会发生基因家族的适应性扩展。 当环境压力源作用于一个物种时就是这种情况。 基因扩增在细菌中更为常见,是一个可逆过程。 基因家族的适应性收缩通常是由功能丧失突变的积累引起的。 过早停止基因转录的无义突变在种群中固定下来,导致基因丢失。 当环境的变化使基因变得多余时,就会发生这个过程。 |
|||
== 功能家族 == |
|||
除了按演化(结构基因家族)分类外,{{le|人类基因组组织基因命名委员会|HUGO Gene Nomenclature Committee}}(HGNC)还在其主干命名法中按功能制作“基因家族”<ref>{{cite web |title=What is a stem symbol? |url=https://www.genenames.org/help/faq/#!/ |website=HGNC FAQ |access-date=2023-05-20 |archive-date=2023-05-20 |archive-url=https://web.archive.org/web/20230520153157/https://www.genenames.org/help/faq/#!/ |dead-url=no }}</ref>。 因此,主干也可以指具有相同功能的基因,通常是同一[[蛋白质复合体]]的一部分。 例如,[[BRCA1]] 和 [[BRCA2]] 是不相关的基因,它们都是根据它们在[[乳腺癌]]中的作用而命名的,而 [[RPS2]] 和 [[RPS3]] 是在同一小亚基中发现的不相关的[[核糖体蛋白]]。 |
|||
HGNC 还维护着一个“基因组”(以前称为“基因家族”)分类。 一个基因可以是多个组的成员,所有组形成一个层次结构。 与主干分类一样,结构组和功能组都存在。 |
|||
== 参见 == |
|||
* {{link-en|基因家族列表|List of gene families}} |
|||
* [[蛋白质家族]] |
|||
==参考资料== |
|||
{{reflist|30em}} |
|||
{{重复序列}} |
|||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
[[bg:Семейство гени]] |
|||
| ⚫ | |||
[[cs:Genová rodina]] |
|||
[[Category:系统发生学]] |
|||
[[en:Gene family]] |
|||
[[es:Familia génica]] |
|||
[[fr:Famille multigénique]] |
|||
[[it:Famiglia genica]] |
|||
[[mk:Генска фамилија]] |
|||
[[nl:Genfamilie]] |
|||
2023年7月18日 (二) 04:30的最新版本
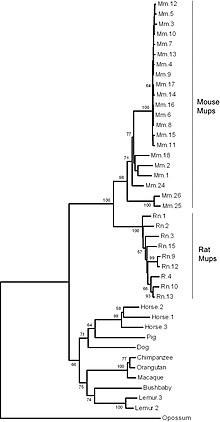
基因家族(英語:Gene family),是来源于同一个祖先,由一个基因通过基因重复而产生两个或更多的拷贝而构成的一组基因,它们在结构和功能上具有明显的相似性,编码相似的蛋白质产物。[1]一个这样的家族是人类血红蛋白亚基的基因,10个基因在不同染色体上的两个簇中,称为α-珠蛋白和β-珠蛋白基因座。这两个基因簇被认为是由于前体基因在大约5亿年前被复制而产生的[2]。
基因根据共享的核苷酸或蛋白质序列被分为家族。 系統發生技术可以用作更严格的测试。外显子在编码序列中的位置可用于推断共同的祖先。 了解基因编码的蛋白质序列可以让研究人员应用方法来发现蛋白质序列之间的相似性,这些相似性提供的信息比DNA序列之间的相似性或差异性更多。
如果一个基因家族的基因编码蛋白质,那么术语蛋白质家族通常以与基因家族类似的方式使用。
基因家族沿特定谱系的扩展或收缩可能是偶然的,也可能是自然选择的结果[3]。区分这两种情况在实践中往往很困难。 最近的工作使用统计模型和算法技术的组合来检测受自然选择影响的基因家族[4]。
国际人类基因组组织(HUGO)的人类基因组组织基因命名委员会(HGNC) 使用基因家族成员的“主干”(或“根”)符号(通过同源性或功能)创建命名方案,并使用分层编号系统来区分各个成员[5][6]。 例如,对于过氧化物氧还酶(Peroxiredoxin)家族,PRDX 是根符号,家族成员为 PRDX1、PRDX2、PRDX3、PRDX4、PRDX5 和 PRDX6。
同一家族基因可以紧密排列在一起,形成一个基因簇,但多数时候,它们是分散在同一染色体的不同位置,或者存在于不同的染色体上的,各自具有不同的表达调控模式。
基本结构
[编辑]
基因组组织的一个层次是将基因分组到几个基因家族中[7][8]。 基因家族是共享共同祖先的相关基因组。 基因家族的成员可能是旁系同源物或直系同源物。 基因旁系同源物是同一物种中具有相似序列的基因,而基因直系同源物是不同物种中具有相似序列的基因。 基因家族在大小、序列多样性和排列方面变化很大。 根据家族内基因的多样性和功能,家族可分为多基因家族或超家族[7][9]。
多基因家族通常由具有相似序列和功能的成员组成,尽管高度差异(在序列和/或功能水平上)不会导致基因从基因家族中移除。 家族中的单个基因可能在同一条染色体上紧密排列在一起,也可能分散在不同染色体上的整个基因组中。 由于它们序列的相似性和重叠功能,家族中的单个基因通常共享调控元件[7][9]。在某些情况下,基因成员具有相同(或几乎相同)的序列。 这些家族允许根据需要在短时间内表达大量基因产物。 其他家族允许在不同细胞类型或生物体发育的不同阶段表达相似但特定的产物[7]。
超家族比单个多基因家族大得多。 超家族包含多达数百个基因,包括多个多基因家族以及单个基因成员。 大量的成员使得超家族可以广泛分散,一些基因聚集在一起,一些基因分散得很远。 这些基因在序列和功能上是多种多样的,显示出不同的表达水平和独立的调节控制[7][9]。
演化
[编辑]基因家族是基因组中信息存储层次结构的一部分,在多细胞生物的演化和多样性中发挥着重要作用。 基因家族是信息和遗传变异的大单位[7]。 随着演化时间的推移,基因家族随着新基因家族的形成和一些基因家族的丢失而扩大和收缩。在几个演化谱系中,基因以相对相同的速率获得和丢失。 当自然选择有利于额外的基因拷贝时,就会发生基因家族的适应性扩展。 当环境压力源作用于一个物种时就是这种情况。 基因扩增在细菌中更为常见,是一个可逆过程。 基因家族的适应性收缩通常是由功能丧失突变的积累引起的。 过早停止基因转录的无义突变在种群中固定下来,导致基因丢失。 当环境的变化使基因变得多余时,就会发生这个过程。
功能家族
[编辑]除了按演化(结构基因家族)分类外,人类基因组组织基因命名委员会(HGNC)还在其主干命名法中按功能制作“基因家族”[10]。 因此,主干也可以指具有相同功能的基因,通常是同一蛋白质复合体的一部分。 例如,BRCA1 和 BRCA2 是不相关的基因,它们都是根据它们在乳腺癌中的作用而命名的,而 RPS2 和 RPS3 是在同一小亚基中发现的不相关的核糖体蛋白。
HGNC 还维护着一个“基因组”(以前称为“基因家族”)分类。 一个基因可以是多个组的成员,所有组形成一个层次结构。 与主干分类一样,结构组和功能组都存在。
参见
[编辑]参考资料
[编辑]- ^ 朱玉贤等 《分子生物学》 高等教育出版社 ISBN 978-7-04-022214-2
- ^ Nussbaum, Robert L.; McInnes, Roderick R.; Willard, Huntington F. Thompson & Thompson Genetics in Medicine 8th. Philadelphia, PA: Elsevier. 2016: 25. ISBN 978-1-4377-0696-3.
- ^ Hartl, D.L. and Clark A.G. 2007. Principles of population genetics. Chapter 7, page 372.
- ^ Demuth, Jeffery P.; Bie, Tijl De; Stajich, Jason E.; Cristianini, Nello; Hahn, Matthew W.; Borevitz, Justin. The Evolution of Mammalian Gene Families. PLOS ONE. 20 December 2006, 1 (1): e85. Bibcode:2006PLoSO...1...85D. PMC 1762380
 . PMID 17183716. doi:10.1371/journal.pone.0000085
. PMID 17183716. doi:10.1371/journal.pone.0000085  .
.
- ^ Daugherty, LC; Seal, RL; Wright, MW; Bruford, EA. Gene family matters: expanding the HGNC resource.. Human Genomics. Jul 5, 2012, 6 (1): 4. PMC 3437568
 . PMID 23245209. doi:10.1186/1479-7364-6-4.
. PMID 23245209. doi:10.1186/1479-7364-6-4.
- ^ HGNC. Gene group help. [2020-10-13]. (原始内容存档于2023-06-01).
- ^ 7.0 7.1 7.2 7.3 7.4 7.5 Hartwell, Leland H.; et al. Genetics : from genes to genomes 4th. New York: McGraw-Hill. 2011. ISBN 978-0073525266.
- ^ Demuth, JP; Hahn, MW. The life and death of gene families.. BioEssays. January 2009, 31 (1): 29–39. PMID 19153999. S2CID 9528185. doi:10.1002/bies.080085.
- ^ 9.0 9.1 9.2 Ohta, Tomoka. Gene families: multigene families and superfamilies. eLS. 2008. ISBN 978-0470015902. doi:10.1038/npg.els.0005126.
- ^ What is a stem symbol?. HGNC FAQ. [2023-05-20]. (原始内容存档于2023-05-20).
